taille <- c(167, 192, 173, 174, 172, 167, 171, 185, 163, 170) # mesures en cm
poids <- c(86, 74, 83, 50, 78, 66, 66, 51, 50, 55) # mesures en Kg
Prog <- c("Bac", "Bac", "Master", "Bac", "Bac", "Master", "Master", NA, "Bac", "Bac") # Programme d'étude
Sexe <- c("H", "H", "F", "H", "H", "H", "F", "H", "H", "H")7 Data frames
En R, les dataframes (ou data frames) sont les structures les plus couramment utilisées pour organiser des jeux de données tabulaires, où chaque ligne représente une observation (ou un individu), et chaque colonne correspond à une caractéristique (ou variable).
Contrairement aux matrices, qui imposent un type unique pour l’ensemble des éléments, les colonnes d’un dataframe peuvent contenir des types de données variés — numériques, textuels, logiques, etc. Cette flexibilité permet de représenter des données réelles de manière plus fidèle, en respectant la nature propre de chaque variable (par exemple : âge en nombre, sexe en facteur, la réussite en booléen (TRUE ou FALSE), etc). C’est ce qui fait des dataframes un outil central en statistique et en science des données.
7.1 Créer un dataframe avec data.frame()
Pour créer un dataframe, on commence par définir les vecteurs qui constitueront les colonnes de notre tableau :
Ensuite, on utilise la fonction data.frame() pour assembler ces vecteurs :
df <- data.frame(Taille = taille, Poids = poids, Prog, Sexe, 11:20)
df Taille Poids Prog Sexe X11.20
1 167 86 Bac H 11
2 192 74 Bac H 12
3 173 83 Master F 13
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 Master H 16
7 171 66 Master F 17
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20Les colonnes d’un dataframe sont systématiquement associées à un nom. Par défaut, R utilise les noms des vecteurs fournis lors de la création (comme pour les colonnes 3 et 4 de df), ou ceux explicitement définis par l’utilisateur (colonnes 1 et 2). En l’absence de nom, R génère automatiquement une étiquette (voir la colonne 5), souvent peu descriptive ou difficile à interpréter. Pour garantir la lisibilité et la clarté des données, il est donc conseillé de nommer manuellement les colonnes sans nom.
En interne, un dataframe est en réalité une liste (dont les éléments correspondent aux colonnes) :
typeof(df)[1] "list"On peut donc accéder aux élements (colonnes) d’un dataframe comme dans une liste :
df[[1]] # Accès à la colonne 1 (résultat = vecteur)
df[["Taille"]] # Idem
df$Taille # Idem
df[1] # Accès à la colonne 1 (résultat = dataframe)
df["Taille"] # Idem [1] 167 192 173 174 172 167 171 185 163 170 Taille
1 167
2 192
3 173
4 174
5 172
6 167
7 171
8 185
9 163
10 170La particularité d’un dataframe entant que liste est que toutes ces élements (colonnes) ont la même longueur, ce qui lui donne une structure bidimensionnelle (lignes × colonnes), similaire à une matrice. On peut donc, par exemple, accéder à une cellule avec la notation df[i, j] :
df[2, 3] # Accède à la 2e ligne, 3e colonne[1] "Bac"De plus, certaines fonctions conçues pour les matrices, comme dim(), rownames(), cbind() et rbind(), par exemple, s’appliquent aussi aux dataframes. Toutefois, contrairement aux matrices qui ne peuvent contenir qu’un seul type de données, les dataframes permettent d’avoir des colonnes de types différents.
Voici la structure ainsi que quelques caractéristiques de notre dataframe df :
str(df)'data.frame': 10 obs. of 5 variables:
$ Taille: num 167 192 173 174 172 167 171 185 163 170
$ Poids : num 86 74 83 50 78 66 66 51 50 55
$ Prog : chr "Bac" "Bac" "Master" "Bac" ...
$ Sexe : chr "H" "H" "F" "H" ...
$ X11.20: int 11 12 13 14 15 16 17 18 19 20length(df) # Nombre de colonnes
names(df) # Noms des colonnes
dim(df) # Dimensions (lignes, colonnes)[1] 5
[1] "Taille" "Poids" "Prog" "Sexe" "X11.20"
[1] 10 5Dans RStudio, on peut explorer un dataframe de manière interactive grâce à la fonction View()
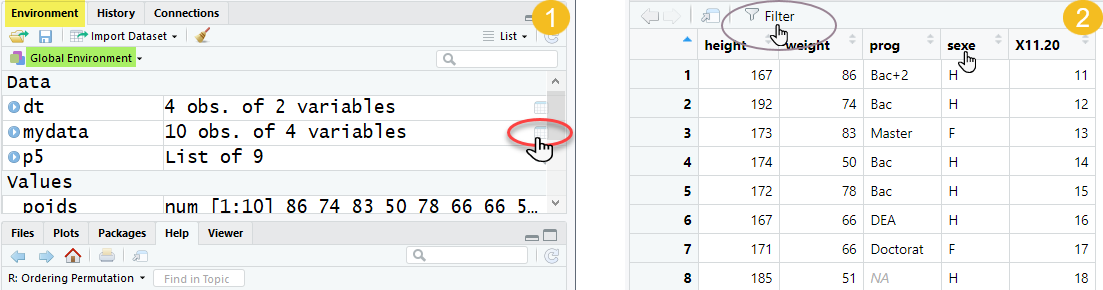
View(df)Ou en cliquant sur l’icône en forme de tableau à droite du nom de l’objet dans l’onglet Environment.
Cela ouvre une interface semblable à une feuille Excel, où on peut faire défiler les lignes, trier les colonnes et inspecter les données facilement. Il y a également un champs de recherche et un bouton “Filter” donnant accès à des options de filtrage.
Avant de poursuivre, il est important de noter que de nombreuses opérations abordées dans les chapitres précédents — qu’elles concernent les vecteurs, les matrices ou les listes — s’appliquent également aux dataframes. Pour éviter les redites, nous allons nous concentrer sur les aspects spécifiques et les fonctionnalités les plus pertinentes propres aux dataframes.
7.2 Extraire des lignes ou des colonnes
Il existe plusieurs méthodes pour accéder à des lignes ou colonnes spécifiques d’un dataframe. Dans cette section, nous allons en explorer quelques-unes à travers des exemples simples.
Parmi ces méthodes, la fonction subset() se distingue par sa clarté et sa facilité d’utilisation. Son syntaxe générale est le suivant :
subset(df, subset, select)df: dataframe à traiter.subset: condition(s) logique(s) pour filtrer les lignes.select: colonnes à conserver (optionnel).
Sa mise en œuvre, appuyée par des exemples concrets, est présentée dans les sections suivantes.
Sélectionner des colonnes
Par numéro/position
Sélectionner une ou plusieurs colonnes
df[c(2, 3)] # Colonnes 2 et 3 df[, c(2, 3)] # Idem df |> _[c(2, 3)] # Idem avec le pipe subset(df, select = c(2, 3)) # Idem avec subset()Output
Poids Prog 1 86 Bac 2 74 Bac 3 83 Master 4 50 Bac 5 78 Bac 6 66 Master 7 66 Master 8 51 <NA> 9 50 Bac 10 55 BacSélectionner tout sauf certaines colonnes
df[-c(2, 3)] subset(de, select = -c(2, 3)) # Idem avec subset()Output
Taille Sexe X11.20 1 167 H 11 2 192 H 12 3 173 F 13 4 174 H 14 5 172 H 15 6 167 H 16 7 171 F 17 8 185 H 18 9 163 H 19 10 170 H 20
Par nom
Sélectionner une colonne
df$Poids # Colonne 'Poids' (résultat = vecteur) df[["Poids"]] # Idem df["Poids"] # Idem (résultat = dataframe) subset(df, select = Poids) # Idem avec subset()Output
[1] 86 74 83 50 78 66 66 51 50 55 Poids 1 86 2 74 3 83 4 50 5 78 6 66 7 66 8 51 9 50 10 55Sélectionner plusieurs colonnes
df[c("Poids", "Prog")] # Deux colonnes df |> _[c("Poids", "Prog")] # Idem subset(df, select = c(Poids, Prog)) # IdemOutput
Poids Prog 1 86 Bac 2 74 Bac 3 83 Master 4 50 Bac 5 78 Bac 6 66 Master 7 66 Master 8 51 <NA> 9 50 Bac 10 55 BacSélectionner tout sauf certaines colonnes
subset(df, select = -c(Poids, Prog))Output
Taille Sexe X11.20 1 167 H 11 2 192 H 12 3 173 F 13 4 174 H 14 5 172 H 15 6 167 H 16 7 171 F 17 8 185 H 18 9 163 H 19 10 170 H 20
Sélectionner des lignes
Par numéro/position
df[c(2, 3), ] # Lignes 2 et 3
df[-c(2, 3), ] # Toutes sauf les lignes 2 et 3
df[c(2, 3), "Poids"] # Lignes 2 et 3, colonne 'Poids'Output
Taille Poids Prog Sexe X11.20
2 192 74 Bac H 12
3 173 83 Master F 13
Taille Poids Prog Sexe X11.20
1 167 86 Bac H 11
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 Master H 16
7 171 66 Master F 17
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20
[1] 74 83Par condition logique (filtrage)
En pratique, il arrive très souvent que l’on souhaite soustraire un sous-ensemble d’observations (lignes) qui remplissent certaines conditions. Voici quelques exemples.
df[df$Sexe == "H", ] # Hommes uniquement
subset(df, subset = Sexe == "H") # IdemOutput
Taille Poids Prog Sexe X11.20
1 167 86 Bac H 11
2 192 74 Bac H 12
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 Master H 16
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20Notez que df[df$Sexe == "H", ], et subset(df, Sexe == "H") donnent le même résultat, mais avec subset(), on peut écrire directement Sexe == "H" sans utiliser df$Sexe, car cette fonction, comme d’autres fonctions en R, applique un mécanisme appelé évaluation non standard (non-standard evaluation). Ce mécanisme implique que la fonction commence par rechercher les noms de variables dans le dataframe fourni, puis, en l’absence de correspondance, les recherche dans l’environnement de travail. À l’inverse, dans une expression comme df[Sexe == "H", ], R applique l’évaluation standard (qui est la règle en général) et cherche tous les symboles, y compris donc df et Sexe, dans l’environnement global. Si aucune variable Sexe n’y existe, l’expression produit une erreur.
On peut aussi combiner plusieurs conditions dans une seule instruction à l’aide de l’opérateur logique & qui signifie “ET”. Cela permet de filtrer les lignes répondant simultanément à plusieurs critères. Exemple :
df[df$Sexe == "H" & df$Poids < mean(df$Poids), ] # Hommes avec poids < moyenne
subset(df, subset = Sexe == "H" & Poids < mean(Poids)) # IdemOutput
Taille Poids Prog Sexe X11.20
4 174 50 Bac H 14
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20On peut également filtrer des lignes et sélectionner des colonnes dans une même instruction :
df |> subset(Sexe == "H" & Poids < mean(Poids)) |> subset(select = c(Taille, Prog))
subset(df, subset = Sexe == "H" & Poids < mean(Poids), select = c(Taille, Prog)) # IdemOutput
Taille Prog
4 174 Bac
8 185 <NA>
9 163 Bac
10 170 Bac7.3 Transformer les colonnes
Dans cette section, nous allons découvrir une fonction très pratique : transform(). Elle permet de modifier, ajouter ou supprimer des colonnes dans un dataframe de manière simple et lisible. Son syntaxe générale est le suivant
transform(df, Newcol1 = expression1, Newcol2 = expression2, ...)df: dataframe à traiter.Newcol1 = expression1: une nouvelle colonne ou une transformation d’une colonne existante.- Plusieurs transformations peuvent être combinées dans une même instruction, séparées par des virgules
Des exemples concrets sont présentés dans les sections suivantes.
Ajouter ou supprimer des variables
Imaginez que nous aimerions ajouter les deux variables suivantes à notre dataframe df :
nom <- c("Benjamin", "Hugo", "Emma", "Alex", "Tom", "Axel", "Alice", "Martin", "Robin", "Enzo")
Grade <- c("A", "A", "C", "B", "B", "B", "C", "A", "A", "A")Pour cela, nous pouvons utiliser l’une des commandes suivantes :
df$Prenom <- nom ; df$Grade <- Grade # Méthode 1 : affectation directe
df <- cbind(df, Prenom = nom, Grade) # Méthode 2 : via cbind
df <- df |> transform(Prenom = nom, Grade = Grade) # Méthode 3 : via transform
df Taille Poids Prog Sexe X11.20 Prenom Grade
1 167 86 Bac H 11 Benjamin A
2 192 74 Bac H 12 Hugo A
3 173 83 Master F 13 Emma C
4 174 50 Bac H 14 Alex B
5 172 78 Bac H 15 Tom B
6 167 66 Master H 16 Axel B
7 171 66 Master F 17 Alice C
8 185 51 <NA> H 18 Martin A
9 163 50 Bac H 19 Robin A
10 170 55 Bac H 20 Enzo AEt voici comment retirer des colonnes d’un dataframe :
df$Prenom <- NULL ; df$X11.20 <- NULL # Méthode 1
df <- df |> transform(Prenom = NULL, X11.20 = NULL) # Méthode 2
df <- df |> subset(selec = -c(Prenom, X11.20)) # Méthode 3
df Taille Poids Prog Sexe Grade
1 167 86 Bac H A
2 192 74 Bac H A
3 173 83 Master F C
4 174 50 Bac H B
5 172 78 Bac H B
6 167 66 Master H B
7 171 66 Master F C
8 185 51 <NA> H A
9 163 50 Bac H A
10 170 55 Bac H AIl est important de noter que le résultat de transform(), comme de subset(), est un dataframe temporaire contenant les modifications demandées. Si vous souhaitez conserver ces dernières, vous devez réassigner le résultat à df (ou à un autre objet).
Transformer des variables existantes
Supposons à présent que nous souhaitons convertir les variables Sexe et Grade en facteurs. Cela peut se faire comme suit :
df$Sexe <- factor(df$Sexe); df$Grade <- factor(df$Grade) # méthode 1
df <- df |> transform(Sexe = factor(Sexe), Grade = factor(Grade)) # méthode 2Nous pouvons vérifier que les colonens/varaibles Sexe et Grade sont effectivement des facteurs en examinant la structure des df.
str(df)'data.frame': 10 obs. of 5 variables:
$ Taille: num 167 192 173 174 172 167 171 185 163 170
$ Poids : num 86 74 83 50 78 66 66 51 50 55
$ Prog : chr "Bac" "Bac" "Master" "Bac" ...
$ Sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2
$ Grade : Factor w/ 3 levels "A","B","C": 1 1 3 2 2 2 3 1 1 1Cela est important, car, comme expliqué précédemment, certaines fonctions appliquent un traitement spécifique aux variables de type facteur. Par exemple, la fonction summary(), utilisée pour calculer des statistiques descriptives, adapte sa sortie en fonction de la nature des variables présentes dans le dataframe. Observez, par exemple, les colonnes “Sexe” et “Grade” ci-dessous :
summary(df) Taille Poids Prog Sexe Grade
Min. :163.0 Min. :50.0 Length:10 F:2 A:5
1st Qu.:167.8 1st Qu.:52.0 Class :character H:8 B:3
Median :171.5 Median :66.0 Mode :character C:2
Mean :173.4 Mean :65.9
3rd Qu.:173.8 3rd Qu.:77.0
Max. :192.0 Max. :86.0 Exemple complet : transformation et création de variables
La fonction transform() est particulièrement utile lorsqu’on souhaite créer de nouvelles variables à partir de celles existantes. Prenons un exemple concret : à partir du dataframe df contenant les variables Taille (en cm) et Poids (en kg), nous souhaitons :
- Créer une nouvelle variable
Taille.mreprésentant la taille en mètres. - Convertir
Poidsen grammes. - Supprimer la variable
Taille. - Calculer l’indice de masse corporelle (IMC), défini par la formule : \text{IMC} = \frac{\text{poids (kg)}}{\text{taille (m)}^2}.
# Méthode 1 : tout en une seule étape
df |> transform(
Taille.m = 0.01 * Taille,
Poids = Poids * 1000,
Taille = NULL,
imc = Poids / (0.01 * Taille)^2
)
# Méthode 2 : en deux étapes successives
df |> transform(
Taille.m = 0.01 * Taille,
Poids = Poids * 1000,
Taille = NULL
) |> transform(
imc = (Poids / 1000) / Taille.m^2
) Poids Prog Sexe Grade Taille.m imc
1 86000 Bac H A 1.67 30.83653
2 74000 Bac H A 1.92 20.07378
3 83000 Master F C 1.73 27.73230
4 50000 Bac H B 1.74 16.51473
5 78000 Bac H B 1.72 26.36560
6 66000 Master H B 1.67 23.66524
7 66000 Master F C 1.71 22.57105
8 51000 <NA> H A 1.85 14.90139
9 50000 Bac H A 1.63 18.81892
10 55000 Bac H A 1.70 19.03114Il est important de comprendre que dans transform(), les variables nouvellement créées ne sont pas immédiatement disponibles : seules celles du dataframe d’origine (fourni) peuvent être utilisées dans la même instruction. Ainsi le code suivant est faux et renvoie une erreur.
df |> transform(
Taille.m = 0.01 * Taille,
Poids = Poids * 1000,
Taille = NULL,
1 imc = (Poids / 1000) / Taille.m^2
)- 1
-
Ici,
Taille.mn’est pas encore reconnu au moment du calcul deimc, car elle vient juste d’être créée dans la même instruction.
Error in eval(substitute(list(...)), `_data`, parent.frame()): object 'Taille.m' not found7.4 Quelques fonctions et manipulations utiles
Cette section présente un ensemble de fonctions pratiques pour manipuler les dataframes en R. Elles de calculer des statistiques, de restructurer les données ou encore de les résumer efficacement.
La fonction with()
Nous avons vu que pour accéder aux variables (ou colonnes) d’un dataframe, on utilise généralement l’opérateur $ ou les crochets. Cependant, cette syntaxe peut devenir verbeuse, notamment dans des expressions complexes ou lorsqu’on utilise des fonctions qui ne prennent pas directement un argument data. C’est dans ce contexte que la fonction with() devient particulièrement utile. Son syntaxe générale est le suivant :
with(df, expression)df: dataframe (ou liste) à traiter.expr: une seule expression ou un bloc d’expressions, regroupées entre accolades{}, que R évaluera dans le contexte du dataframe (mécanisme d’évaluation non standard).
Par exemple, imaginez que l’on souhaite calculer le coefficient de variation (sd/mean) de Poids/Taille. On peut taper
sd(df$Poids / df$Taille) / mean(df$Poids / df$Taille)[1] 0.2177457Voici une syntaxe alternative, plus lisible, grâce à l’utilisation de la fonction with() :
with(df, sd(Poids / Taille) / mean(Poids / Taille))
# ou
df |> with({
ratio <- Poids / Taille
mean(ratio) / sd(ratio)
})[1] 0.2177457Dans le dernier code ci-dessus :
- Le pipe
|>transmet l’objetdf, comme premier argument, à la fonctionwith(). - Grâce à
with(), on peut utiliser directement les noms des colonnes (Poids,Taille) sans avoir à écriredf$à chaque fois. - Les accolades
{}permettent de regrouper plusieurs instructions dans un bloc d’expression. Sans elles,with()ne pourrait exécuter qu’une seule instruction.
Voici un autre exemple où l’on utilise la fonction with() pour extraire les hommes dont le poids est inférieur au poids moyen.
with(df, df[Sexe == "H" & Poids < mean(Poids), ]) Taille Poids Prog Sexe Grade
4 174 50 Bac H B
8 185 51 <NA> H A
9 163 50 Bac H A
10 170 55 Bac H AObtenir un aperçu ou un résumé
head(df) # Les six premières lignes
tail(df) # Les six dernières lignes
summary(df) # Statistiques descriptives par colonneOutput
Taille Poids Prog Sexe Grade
1 167 86 Bac H A
2 192 74 Bac H A
3 173 83 Master F C
4 174 50 Bac H B
5 172 78 Bac H B
6 167 66 Master H B
Taille Poids Prog Sexe Grade
5 172 78 Bac H B
6 167 66 Master H B
7 171 66 Master F C
8 185 51 <NA> H A
9 163 50 Bac H A
10 170 55 Bac H A
Taille Poids Prog Sexe Grade
Min. :163.0 Min. :50.0 Length:10 F:2 A:5
1st Qu.:167.8 1st Qu.:52.0 Class :character H:8 B:3
Median :171.5 Median :66.0 Mode :character C:2
Mean :173.4 Mean :65.9
3rd Qu.:173.8 3rd Qu.:77.0
Max. :192.0 Max. :86.0 Statistiques par groupe avec aggregate()
Il arrive souvent qu’on veuille calculer des statistiques descriptives tels que le minimum, la moyenne, la médiane, d’une variable pour un sous-ensemble (regroupement ) d’individus d’un jeu de données. Cela est possible garce à la fonction aggregate(). Nous allons parler ici de la version “formula” dont le syntaxe (de base) est donné par
aggregate(formula, data, FUN, ...)où formula est une formule de type y ~ x, dans laquelle y représente la ou les variables d’intérêt, et x désigne le ou les facteurs de regroupement (variables de découpage). data est le dataframe contenant les données, et FUN est la fonction à appliquer (ex. mean, sum, quantile, etc.). Les ... permettent, si nécessaire, de passer des paramètres supplémentaires à la fonction FUN. Il est fort conseillé, dans ce cas, de nommer explicitement ces arguments. Voici quelques exemples.
# Moyenne de Taille pour chaque modalité de Sexe
aggregate(Taille ~ Sexe, data = df, FUN = mean)
# Quantiles de Taille pour chaque combinaison de Sexe et Grade
aggregate(Taille ~ Sexe + Grade, data = df, FUN = quantile)
# Médiane (quantile à 0.5) de Taille pour chaque modalité de Sexe
aggregate(Taille ~ Sexe, data = df, FUN = quantile, probs = 0.5)
# Médiane de Taille et de Poids pour chaque combinaison de Sexe et Grade.
aggregate(cbind(Taille, Poids) ~ Sexe + Grade, data = df, FUN = quantile, probs = 0.5)Output
Sexe Taille
1 F 172.00
2 H 173.75
Sexe Grade Taille.0% Taille.25% Taille.50% Taille.75% Taille.100%
1 H A 163.0 167.0 170.0 185.0 192.0
2 H B 167.0 169.5 172.0 173.0 174.0
3 F C 171.0 171.5 172.0 172.5 173.0
Sexe Taille
1 F 172
2 H 171
Sexe Grade Taille Poids
1 H A 170 55.0
2 H B 172 66.0
3 F C 172 74.5Dénombrer avec table() et xtabs()
La fonction table() est utile pour résumer et analyser des données catégorielles. Elle permet de créer des tableaux de fréquences, càd de compter le nombre d’occurrences de chaque modalité d’une ou plusieurs variables.
# Tableau simple (une variable)
table(df$Sexe)
# Tableau croisé (deux variables)
table(df$Grade, df$Sexe) # ou df |> with(table(Grade, Sexe))Output
F H
2 8
F H
A 0 5
B 0 3
C 2 0À la place de table(), on peut utiliser la fonction xtabs(), qui offre une plus grande flexibilité avec les dataframes grâce à sa syntaxe basée sur les formules.
xtabs(~Sexe, data = df)
xtabs(~ Grade + Sexe, data = df)Output
Sexe
F H
2 8
Sexe
Grade F H
A 0 5
B 0 3
C 2 0Renommer des lignes ou colonnes
names(df)[c(3, 5)] <- c("Program", "id") # Renommer des colonnes
rownames(df) <- paste0(1:10, "th") # Renommer les lignes
df Taille Poids Program Sexe id
1th 167 86 Bac H A
2th 192 74 Bac H A
3th 173 83 Master F C
4th 174 50 Bac H B
5th 172 78 Bac H B
6th 167 66 Master H B
7th 171 66 Master F C
8th 185 51 <NA> H A
9th 163 50 Bac H A
10th 170 55 Bac H AOrdonner les lignes
df[order(df$Taille), ] # Tri croissant par Taille
df[order(-df$Taille), ] # Tri décroissant par Taille
df[order(df$Taille, df$Poids), ] # Tri par Taille puis PoidsOutput
Taille Poids Program Sexe id
9th 163 50 Bac H A
1th 167 86 Bac H A
6th 167 66 Master H B
10th 170 55 Bac H A
7th 171 66 Master F C
5th 172 78 Bac H B
3th 173 83 Master F C
4th 174 50 Bac H B
8th 185 51 <NA> H A
2th 192 74 Bac H A
Taille Poids Program Sexe id
2th 192 74 Bac H A
8th 185 51 <NA> H A
4th 174 50 Bac H B
3th 173 83 Master F C
5th 172 78 Bac H B
7th 171 66 Master F C
10th 170 55 Bac H A
1th 167 86 Bac H A
6th 167 66 Master H B
9th 163 50 Bac H A
Taille Poids Program Sexe id
9th 163 50 Bac H A
6th 167 66 Master H B
1th 167 86 Bac H A
10th 170 55 Bac H A
7th 171 66 Master F C
5th 172 78 Bac H B
3th 173 83 Master F C
4th 174 50 Bac H B
8th 185 51 <NA> H A
2th 192 74 Bac H AEmpiler des collones avec stack()
Empiler des colonnes consiste à transformer plusieurs colonnes d’un dataframe en une seule colonne. Cette opération est particulièrement utile pour restructurer les données dans un format plus adapté à certaines tâches, comme la création de graphiques.
La fonction stack() permet d’empiler facilement les colonnes d’un dataframe, comme illustré dans l’exemple suivant.
df <- data.frame(
Nom = c("Alice", "Bob", "Charlie"),
Math = c(12, 15, 14),
Physique = c(13, 14, 16),
Chimie = c(11, 13, 15)
)
df Nom Math Physique Chimie
1 Alice 12 13 11
2 Bob 15 14 13
3 Charlie 14 16 15stack(df) values ind
1 Alice Nom
2 Bob Nom
3 Charlie Nom
4 12 Math
5 15 Math
6 14 Math
7 13 Physique
8 14 Physique
9 16 Physique
10 11 Chimie
11 13 Chimie
12 15 ChimieDans cette sortie,
- La colonne
valuescontient toutes les valeurs des colonnes empilées. - La colonne
indindique de quelle colonne chaque valeur provient.
Il est possible d’empiler uniquement certaines colonnes en utilisant l’argument select :
stack(df, select = c(Math, Physique, Chimie)) values ind
1 12 Math
2 15 Math
3 14 Math
4 13 Physique
5 14 Physique
6 16 Physique
7 11 Chimie
8 13 Chimie
9 15 ChimieOn peut regrouper les colonnes empilées (“Math”, “Physique”, “Chimie”) avec les autres (“Nom”) dans un seul dataframe, ce qui rend les données plus lisibles.
cbind(
subset(df, select = -c(Math, Physique, Chimie)), # Partie non empilée
stack(df, select = c(Math, Physique, Chimie)) # Partie empilée
) Nom values ind
1 Alice 12 Math
2 Bob 15 Math
3 Charlie 14 Math
4 Alice 13 Physique
5 Bob 14 Physique
6 Charlie 16 Physique
7 Alice 11 Chimie
8 Bob 13 Chimie
9 Charlie 15 Chimie7.5 Variantes de dataframes
Bien que les dataframes de base soient extrêmement utiles, la communauté R a développé, au fil du temps, des alternatives qui en améliorent les fonctionnalités dans des contextes spécifiques. Deux variantes particulièrement populaires sont les tibbles et data tables.
Dans cette section, nous allons aborder uniquement les tibbles. Nous reviendrons sur les data tables plus loin.
Un tibble est une réinvention moderne du dataframe, développée dans le cadre de l’écosystème tidyverse. Il conserve toutes les fonctionnalités des dataframes tout en offrant un comportement amélioré et plus cohérent.
La création d’un tibble est similaire à celle d’un dataframe.
taille <- c(167, 192, 173, 174, 172, 167, 171, 185, 163, 170)
poids <- c(86, 74, 83, 50, 78, 66, 66, 51, 50, 55)
Prog <- c("Bac", "Bac", "Master", "Bac", "Bac", "Master", "Master", NA, "Bac", "Bac")
Sexe <- c("H", "H", "F", "H", "H", "H", "F", "H", "H", "H") |> factor()require(tibble) # <-- Au préalable install.packages("tibble")tb <- tibble(Taille = taille, Poids = poids, Prog, Sexe)
tb# A tibble: 10 × 4
Taille Poids Prog Sexe
<dbl> <dbl> <chr> <fct>
1 167 86 Bac H
2 192 74 Bac H
3 173 83 Master F
4 174 50 Bac H
5 172 78 Bac H
6 167 66 Master H
7 171 66 Master F
8 185 51 <NA> H
9 163 50 Bac H
10 170 55 Bac H L’affichage des tibbles est aussi similaire à celui des dataframes avec toutefois quelques particularités, notamment :
- La première ligne montre la nature de l’objet (tibble) et ces dimensions (ici 10 x 4, pour 10 lignes et 4 colonnes).
- Le type de chaque variable visible est décrit, en abrégé, juste après les noms de colonnes.
- Par défaut, les tibbles affichent uniquement les 10 premières lignes et autant de colonnes que la largeur de la console le permet, avec un message indiquant les lignes ou colonnes non affichées.
Notez qu’il est possible de créer un tible ligne par ligne à l’aide de la fonction tribble().
tribble(
~Taille, ~Poids, ~Prog, ~Sexe,
67, 86, "Bac+2", "H",
192, 74, "Bac", "H",
173, 83, "Master", "F"
)# A tibble: 3 × 4
Taille Poids Prog Sexe
<dbl> <dbl> <chr> <chr>
1 67 86 Bac+2 H
2 192 74 Bac H
3 173 83 Master F Malgré les différences signalées ci-dessus, un tibble et un dataframe se manipulent exactement de la même manière. On peut donc appliquer sur un tibble tout ce que l’on a appris sur les dataframes. Il est par ailleurs possible de transformer un tibble en dataframe, et vice versa.
df <- as.data.frame(tb)
str(df)'data.frame': 10 obs. of 4 variables:
$ Taille: num 167 192 173 174 172 167 171 185 163 170
$ Poids : num 86 74 83 50 78 66 66 51 50 55
$ Prog : chr "Bac" "Bac" "Master" "Bac" ...
$ Sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2tb <- as_tibble(df)
str(tb)tibble [10 × 4] (S3: tbl_df/tbl/data.frame)
$ Taille: num [1:10] 167 192 173 174 172 167 171 185 163 170
$ Poids : num [1:10] 86 74 83 50 78 66 66 51 50 55
$ Prog : chr [1:10] "Bac" "Bac" "Master" "Bac" ...
$ Sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2