7 Les data frames
En statistique et en science des données, il est recommandé que chaque jeu de données (ou dataset en anglais) soit structuré sous forme de tableau, où chaque ligne représente une observation (ou un individu), et chaque colonne correspond à une caractéristique (ou variable).
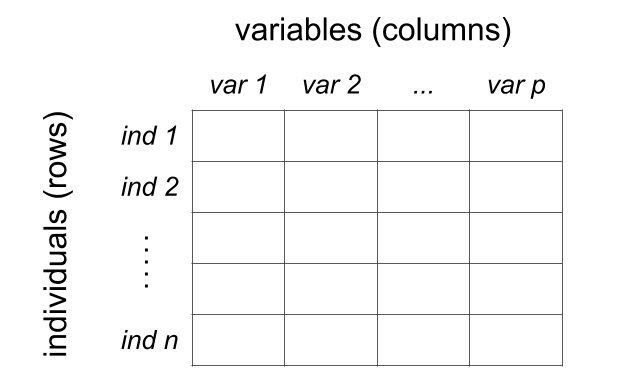
En R, les data frames (ou dataframes) sont les instruments les plus couramment utilisés pour stocker ce type d’objets. Les colonnes d’un dataframe peuvent contenir des types de données différents (numériques, caractères, logiques, etc.), ce qui offre plus de flexibilité que les matrices. Cette capacité à mélanger les types permet de représenter des données réelles de manière plus fidèle, où chaque variable peut avoir sa propre nature.
Pour créer un dataframe, nous allons commencer par créer les vecteurs qui constitueront les colonnes de notre future dataframe.
taille <- c(167, 192, 173, 174, 172, 167, 171, 185, 163, 170) # mesures en cm
poids <- c(86, 74, 83, 50, 78, 66, 66, 51, 50, 55) # mesures en Kg
prog <- c("Bac", "Bac", "Master", "Bac", "Bac", "Master", "Master", NA, "Bac", "Bac") # programme d'étude
sexe <- c("H", "H", "F", "H", "H", "H", "F", "H", "H", "H")Maintenant il suffit d’utiliser la fonction data.frame() :
height weight prog sexe X11.20
1 167 86 Bac H 11
2 192 74 Bac H 12
3 173 83 Master F 13
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 Master H 16
7 171 66 Master F 17
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20Les colonnes d’un dataframe sont toujours nommées. Ils héritent automatiquement les noms des vecteurs fournis (voir la 3e et la 4e colonne de df). Au souhait, l’utilisateur peut attribuer d’autres noms (voir la 1re et la 2e colonne). Dans le cas où un vecteur n’a pas de nom, R attribue lui même un nom (voir la 5e colonne). Il est conseillé de ne pas laisser R faire, car cela résulte souvent en des noms non pertinents.
Les lignes quant à elles sont automatiquement nommées/numérotées par ordre (1 = première ligne, 2 = deuxième ligne,…). En pratique, on a rarement besoin de modifier ces noms.
En interne, un dataframe est une liste dont les éléments sont les colonnes, auxquelles on peut accéder, comme on le ferait avec une liste, en utilisant dataframe$colonne.
[1] "list" [1] 167 192 173 174 172 167 171 185 163 170La particularité d’un dataframe est que toutes ses colonnes ont la même taille, ce qui lui confère une structure bidimensionnelle composée de lignes et de colonnes. Cette organisation, similaire à celle d’une matrice, facilite l’accès aux données, notamment à l’aide de df[i, j] :
[1] "Bac"De plus, certaines fonctions conçues pour les matrices, comme dim(), rownames(), cbind() et rbind(), par exemple, s’appliquent aussi aux dataframes. Toutefois, contrairement aux matrices qui ne peuvent contenir qu’un seul type de données, les dataframes permettent d’avoir des colonnes de types différents.
Voici la structure et quelques caractéristiques de notre dataframe df :
'data.frame': 10 obs. of 5 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac" "Bac" "Master" "Bac" ...
$ sexe : chr "H" "H" "F" "H" ...
$ X11.20: int 11 12 13 14 15 16 17 18 19 20[1] 5
[1] "height" "weight" "prog" "sexe" "X11.20"
[1] 10 5Dans RStudio, on peut explorer un dataframe de manière interactive grâce à la fonction View()
ou en cliquanty sur l’icône en forme de tableau située à droite du nom de l’objet dans l’onglet Environment (en haut à droite).
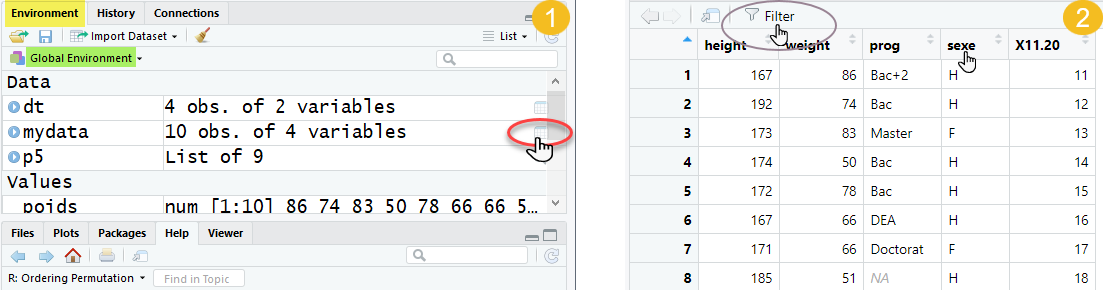
Cela ouvre une interface semblable à une feuille Excel, où on peut faire défiler les lignes, trier les colonnes et inspecter les données facilement. Il y a également un champs de recherche et un bouton “Filter” donnant accès à des options de filtrage.
Avant de poursuivre, il est important de noter que de nombreuses opérations abordées dans les chapitres précédents — qu’elles concernent les vecteurs, les matrices ou les listes — s’appliquent également aux dataframes. Afin d’éviter les répétitions, nous allons désormais nous concentrer sur les aspects spécifiques et les fonctionnalités les plus pertinentes propres aux DataFrames.
7.1 Accéder aux éléments d’un dataframe
Il existe plusieurs méthodes pour accéder aux éléments d’un dataframe en R, chacune adaptée à des besoins spécifiques. Dans cette section, nous allons en explorer quelques-unes à travers des exemples simples.
Parmi ces méthodes, la fonction subset() se distingue par sa simplicité et son intuitivité. Son syntaxe générale est le suivant :
df: dataframe à traiter.subset: condition(s) logique(s) pour filtrer les lignes.select: colonnes à conserver (optionnel).
Plus de détails sont présentés ci-après.
Sélectionner certaines colonnes
+ Par numéro/position
# Sélectionner une ou plusieurs colonnes
df[, c(2, 3)]
df[c(2, 3)] # idem
df |> _[c(2, 3)] # idem (avec le pipe)
df |> subset(select = c(2, 3)) # idem (avec subset) weight prog
1 86 Bac
2 74 Bac
3 83 Master
4 50 Bac
5 78 Bac
6 66 Master
7 66 Master
8 51 <NA>
9 50 Bac
10 55 Bac height sexe X11.20
1 167 H 11
2 192 H 12
3 173 F 13
4 174 H 14
5 172 H 15
6 167 H 16
7 171 F 17
8 185 H 18
9 163 H 19
10 170 H 20+ Par nom
# Sélectionner une colonne
df$weight # output --> vecteur
df |> _$weight # idem
df["weight"] # output --> dataframe
df |> subset(select = weight) # idem weight
1 86
2 74
3 83
4 50
5 78
6 66
7 66
8 51
9 50
10 55# Sélectionner plusieurs colonnes
df[c("weight", "prog")]
df |> _[c("weight", "prog")]
df |> subset(select = c(weight, prog)) weight prog
1 86 Bac
2 74 Bac
3 83 Master
4 50 Bac
5 78 Bac
6 66 Master
7 66 Master
8 51 <NA>
9 50 Bac
10 55 Bac height sexe X11.20
1 167 H 11
2 192 H 12
3 173 F 13
4 174 H 14
5 172 H 15
6 167 H 16
7 171 F 17
8 185 H 18
9 163 H 19
10 170 H 20Sélectionner certaines lignes
+ Par numéro/position
height weight prog sexe X11.20
2 192 74 Bac H 12
3 173 83 Master F 13 height weight prog sexe X11.20
1 167 86 Bac H 11
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 Master H 16
7 171 66 Master F 17
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20[1] 74 83+ Par condition logique (filtrage)
En pratique, il arrive très souvent que l’on souhaite soustraire un sous-ensemble d’observations (lignes) qui remplissent certaines conditions. Voici quelques exemples.
df[df$sexe == "H", ] # sélectionner uniquement les hommes
subset(df, subset = sexe == "H") # idem
df |> subset(sexe == "H") # idem height weight prog sexe X11.20
1 167 86 Bac H 11
2 192 74 Bac H 12
4 174 50 Bac H 14
5 172 78 Bac H 15
6 167 66 Master H 16
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20# les hommes dont le poids est inférieur au poids moyen
df[df$sexe == "H" & df$weight < mean(df$weight), ]
df |> subset(sexe == "H" & weight < mean(weight)) # idem height weight prog sexe X11.20
4 174 50 Bac H 14
8 185 51 <NA> H 18
9 163 50 Bac H 19
10 170 55 Bac H 20# idem, mais en plus nous conservons uniquement les colonnes "height" et "prog"
df |> subset(sexe == "H" & weight < mean(weight), select = c(height, prog)) height prog
4 174 Bac
8 185 <NA>
9 163 Bac
10 170 Bac7.2 Modifier les colonnes d’un dataframe
Dans cette section, nous allons découvrir une fonction très pratique : transform(). Elle permet de modifier, ajouter ou supprimer des colonnes dans un dataframe de manière simple et lisible. Son syntaxe générale est le suivant
df: dataframe à traiter.Newcol1 = expression1: une nouvelle colonne ou une transformation d’une colonne existante.- On peut ajouter autant de transformations que nécessaire, séparées par des virgules.
Plus de détails sont présentés ci-après.
Ajouter des variables (colonnes)
Pour commencer, imaginez que nous aimerions ajouter les deux variables suivantes à notre dataframe df .
nom <- c("Benjamin", "Hugo", "Emma", "Alex", "Tom", "Axel", "Alice", "Martin", "Robin", "Enzo")
grade <- c("A", "A", "C", "B", "B", "B", "C", "A", "A", "A")Pour cela, nous pouvons utiliser l’une des commandes suivantes :
df$prenom <- nom ; df$grade <- grade # méthode 1
df <- cbind(df, prenom = nom, grade) # méthode 2
df <- df |> transform(prenom = nom, grade = grade) # méthode 3 height weight prog sexe X11.20 prenom grade
1 167 86 Bac H 11 Benjamin A
2 192 74 Bac H 12 Hugo A
3 173 83 Master F 13 Emma C
4 174 50 Bac H 14 Alex B
5 172 78 Bac H 15 Tom B
6 167 66 Master H 16 Axel B
7 171 66 Master F 17 Alice C
8 185 51 <NA> H 18 Martin A
9 163 50 Bac H 19 Robin A
10 170 55 Bac H 20 Enzo ASupprimer des variables
Et voilà comme supprimer des variables :
df$prenom <- NULL ; df$X11.20 <- NULL # méthode 1
df <- df |> transform(prenom = NULL, X11.20 = NULL) # méthode 2
df <- df |> subset(selec = -c(prenom, X11.20)) # méthode 3 height weight prog sexe grade
1 167 86 Bac H A
2 192 74 Bac H A
3 173 83 Master F C
4 174 50 Bac H B
5 172 78 Bac H B
6 167 66 Master H B
7 171 66 Master F C
8 185 51 <NA> H A
9 163 50 Bac H A
10 170 55 Bac H ATransformer des variables existantes
Supposons maintenant que nous voulons transformer les variables sexe et grade en facteur. Cela peut se faire comme ceci
df$sexe <- factor(df$sexe); df$grade <- factor(df$grade) # méthode 1
df <- df |> transform(sexe = factor(sexe), grade = factor(grade)) # méthode 2Nous pouvons vérifier que les colonens/varaibles sexe et grade sont effectivement des facteurs en examinant la structure des df.
'data.frame': 10 obs. of 5 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac" "Bac" "Master" "Bac" ...
$ sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2
$ grade : Factor w/ 3 levels "A","B","C": 1 1 3 2 2 2 3 1 1 1Cela est important, car, comme expliqué précédemment, certaines fonctions appliquent un traitement spécifique aux variables de type facteur. Par exemple, la fonction summary(), utilisée pour calculer des statistiques descriptives, adapte sa sortie en fonction de la nature des variables présentes dans le dataframe. Observez, par exemple, les colonnes “sexe” et “grade” ci-dessous :
height weight prog sexe grade
Min. :163 Min. :50.0 Length:10 F:2 A:5
1st Qu.:168 1st Qu.:52.0 Class :character H:8 B:3
Median :172 Median :66.0 Mode :character C:2
Mean :173 Mean :65.9
3rd Qu.:174 3rd Qu.:77.0
Max. :192 Max. :86.0 La fonction transform() est particulièrement utile lorsqu’on souhaite créer de nouvelles variables à partir de celles existantes. Prenons un exemple concret : à partir du dataframe df contenant les variables height (en cm) et weight (en kg), nous souhaitons :
- créer une nouvelle variable
height.mreprésentant la taille en mètres. - convertir
weighten grammes. - supprimer la variable
height. - calculer l’indice de masse corporelle (IMC), défini par la formule : \(\text{IMC} = \frac{\text{poids (kg)}}{\text{taille (m)}^2}\).
# méthode 1 : tout en une seule étape
df |> transform(
height.m = 0.01 * height,
weight = weight * 1000,
height = NULL,
imc = weight / (0.01 * height)^2
)
# méthode 2 : en deux étapes successives
df |> transform(
height.m = 0.01 * height,
weight = weight * 1000,
height = NULL
) |> transform(
imc = (weight / 1000) / height.m^2
) weight prog sexe grade height.m imc
1 86000 Bac H A 1.67 30.8
2 74000 Bac H A 1.92 20.1
3 83000 Master F C 1.73 27.7
4 50000 Bac H B 1.74 16.5
5 78000 Bac H B 1.72 26.4
6 66000 Master H B 1.67 23.7
7 66000 Master F C 1.71 22.6
8 51000 <NA> H A 1.85 14.9
9 50000 Bac H A 1.63 18.8
10 55000 Bac H A 1.70 19.0Il est important de comprendre que dans transform(), les variables créées ne sont pas immédiatement disponibles : seules celles du dataframe d’origine (fourni) peuvent être utilisées dans la même instruction. Ainsi le code suivant est faux et renvoie une erreur.
df |> transform(
height.m = 0.01 * height,
weight = weight * 1000,
height = NULL,
imc = (weight / 1000) / height.m^2
)Error in eval(substitute(list(...)), `_data`, parent.frame()): object 'height.m' not foundDans ce cas, height.m n’est pas encore reconnu au moment du calcul de imc, car elle vient juste d’être créée dans la même instruction.
Notez aussi que les commandes ci-dessus ne modifient pas le dataframe df lui-même. Le résultat de transform(), comme de subset(), est un nouveau dataframe, temporaire, qui contient les modifications demandées. Si vous souhaitez conserver ces dernières, vous devez réassigner le résultat à df ou à un autre objet :
7.3 Quelques fonctions et manipulations utiles
+ La fonction with()
Nous avons vu que pour accéder aux variables (ou colonnes) d’un dataframe, on utilise généralement l’opérateur $ ou les crochets. Cependant, cette syntaxe peut devenir verbeuse, notamment dans des expressions complexes ou lorsqu’on utilise des fonctions qui ne prennent pas directement un argument data. C’est dans ce contexte que la fonction with() devient particulièrement utile. Son syntaxe générale est le suivant :
df: dataframe (ou liste) à traiter.expr: une seule expression ou un bloc d’expressions, regroupées entre accolades{}, que R évaluera dans le contexte du dataframe.
Par exemple, imaginez que l’on souhaite calculer la moyenne de weight/height. On peut taper
[1] 0.381Voici une syntaxe alternative, plus lisible, grâce à l’utilisation0 de la fonction with() :
[1] 0.381Dans le dernier code ci-dessus :
- Le pipe
|>transmet l’objetdf, comme premier argument, à la fonctionwith(). - Grâce à
with(), on peut utiliser directement les noms des colonnes (weight,height) sans avoir à écriredf$à chaque fois. - Les accolades
{}permettent de regrouper plusieurs instructions dans un bloc d’expression. Sans elles,with()ne pourrait exécuter qu’une seule instruction.
Voici un autre exemple où l’on utilise la fonction with() pour extraire les hommes dont le poids est inférieur au poids moyen.
height weight prog sexe grade
4 174 50 Bac H B
8 185 51 <NA> H A
9 163 50 Bac H A
10 170 55 Bac H A+ Obtenir un aperçu/résumé
height weight prog sexe grade
1 167 86 Bac H A
2 192 74 Bac H A
3 173 83 Master F C
4 174 50 Bac H B
5 172 78 Bac H B
6 167 66 Master H B height weight prog sexe grade
5 172 78 Bac H B
6 167 66 Master H B
7 171 66 Master F C
8 185 51 <NA> H A
9 163 50 Bac H A
10 170 55 Bac H A height weight prog sexe grade
Min. :163 Min. :50.0 Length:10 F:2 A:5
1st Qu.:168 1st Qu.:52.0 Class :character H:8 B:3
Median :172 Median :66.0 Mode :character C:2
Mean :173 Mean :65.9
3rd Qu.:174 3rd Qu.:77.0
Max. :192 Max. :86.0 + Statistiques calculées par groupe
Il arrive souvent qu’on veuille calculer des statistiques descriptives tels que le minimum, la moyenne, la médiane, d’une variable pour un sous-ensemble (regroupement ) d’individus d’un jeu de données. Cela est possible garce à la fonction aggregate(). Nous allons parler ici de la version “formula” dont le syntaxe (de base) est donné par
où formula est une formule de type y ~ x, où y est la variable d’intérêt (variable à résumer) et x le facteur de découpage (variable de groupe), data est le dataframe contenant les données, et FUN est la fonction à appliquer (ex. mean, sum, quantile, etc.). Les ... permettent, si nécessaire, de passer des paramètres supplémentaires à la fonction FUN. Il est fort conseillé, dans ce cas, de nommer explicitement ces arguments. Voici quelques exemples.
sexe height
1 F 172
2 H 174# quantiles de la taille par sexe et grade
aggregate(height ~ sexe + grade, data = df, FUN = quantile) sexe grade height.0% height.25% height.50% height.75% height.100%
1 H A 163 167 170 185 192
2 H B 167 170 172 173 174
3 F C 171 172 172 172 173 sexe height
1 F 172
2 H 171# médiane de la taille et le poids par sexe et grade
aggregate(cbind(height, weight) ~ sexe + grade, data = df, FUN = quantile, probs = 0.5) sexe grade height weight
1 H A 170 55.0
2 H B 172 66.0
3 F C 172 74.5+ Dénombrer les individus
La fonction table() est utile pour résumer et analyser des données catégorielles. Elle permet de créer des tableaux de fréquences, càd de compter le nombre d’occurrences de chaque modalité d’une ou plusieurs variables.
F H
2 8
F H
A 0 5
B 0 3
C 2 0À la place de table(), on peut utiliser la fonction xtabs(), qui offre une plus grande flexibilité avec les dataframes grâce à sa syntaxe basée sur les formules.
sexe
F H
2 8 sexe
grade F H
A 0 5
B 0 3
C 2 0+ Renommer des ligens/colonnes
height weight program sexe id
1th 167 86 Bac H A
2th 192 74 Bac H A
3th 173 83 Master F C
4th 174 50 Bac H B
5th 172 78 Bac H B
6th 167 66 Master H B
7th 171 66 Master F C
8th 185 51 <NA> H A
9th 163 50 Bac H A
10th 170 55 Bac H A+ Ordonner les lignes
height weight program sexe id
9th 163 50 Bac H A
1th 167 86 Bac H A
6th 167 66 Master H B
10th 170 55 Bac H A
7th 171 66 Master F C
5th 172 78 Bac H B
3th 173 83 Master F C
4th 174 50 Bac H B
8th 185 51 <NA> H A
2th 192 74 Bac H A height weight program sexe id
2th 192 74 Bac H A
8th 185 51 <NA> H A
4th 174 50 Bac H B
3th 173 83 Master F C
5th 172 78 Bac H B
7th 171 66 Master F C
10th 170 55 Bac H A
1th 167 86 Bac H A
6th 167 66 Master H B
9th 163 50 Bac H A height weight program sexe id
9th 163 50 Bac H A
6th 167 66 Master H B
1th 167 86 Bac H A
10th 170 55 Bac H A
7th 171 66 Master F C
5th 172 78 Bac H B
3th 173 83 Master F C
4th 174 50 Bac H B
8th 185 51 <NA> H A
2th 192 74 Bac H A+ Empiler des collones
Empiler des colonnes consiste à transformer plusieurs colonnes d’un dataframe en une seule colonne. Cette opération est particulièrement utile pour restructurer les données dans un format plus adapté à certaines tâches, comme la création de graphiques.
La fonction stack() permet d’empiler facilement les colonnes d’un dataframe, comme illustré dans l’exemple suivant.
df <- data.frame(
Nom = c("Alice", "Bob", "Charlie"),
Math = c(12, 15, 14),
Physique = c(13, 14, 16),
Chimie = c(11, 13, 15)
)
df Nom Math Physique Chimie
1 Alice 12 13 11
2 Bob 15 14 13
3 Charlie 14 16 15 values ind
1 Alice Nom
2 Bob Nom
3 Charlie Nom
4 12 Math
5 15 Math
6 14 Math
7 13 Physique
8 14 Physique
9 16 Physique
10 11 Chimie
11 13 Chimie
12 15 ChimieDans cette sortie,
- La colonne
valuescontient toutes les valeurs des colonnes empilées. - La colonne
indindique de quelle colonne chaque valeur provient.
Il est possible d’empiler uniquement certaines colonnes en utilisant l’argument select :
values ind
1 12 Math
2 15 Math
3 14 Math
4 13 Physique
5 14 Physique
6 16 Physique
7 11 Chimie
8 13 Chimie
9 15 ChimieOn peut regrouper les colonnes empilées (“Math”, “Physique”, “Chimie”) avec les autres (“Nom”) dans un seul dataframe, ce qui rend les données plus lisibles.
cbind(
subset(df, select = -c(Math, Physique, Chimie)), # partie non-empilée
stack(df, select = c(Math, Physique, Chimie)) # partie empilée
) Nom values ind
1 Alice 12 Math
2 Bob 15 Math
3 Charlie 14 Math
4 Alice 13 Physique
5 Bob 14 Physique
6 Charlie 16 Physique
7 Alice 11 Chimie
8 Bob 13 Chimie
9 Charlie 15 Chimie7.4 Variantes de dataframes
Bien que les dataframes de base soit extrêmement utile, la communauté R a développé au cours de temps des alternatives qui améliorent ses fonctionnalités dans des domaines spécifiques. Deux variantes particulièrement populaires sont les tibbles et data tables.
7.4.1 Tibbles
Un tibble est une réinvention moderne du dataframe, développée dans le cadre de l’écosystème tidyverse. Il conserve toutes les fonctionnalités des dataframes tout en offrant un comportement amélioré et plus cohérent.
La création d’un tibble est similaire à celle d’un dataframe.
taille <- c(167, 192, 173, 174, 172, 167, 171, 185, 163, 170)
poids <- c(86, 74, 83, 50, 78, 66, 66, 51, 50, 55)
prog <- c("Bac", "Bac", "Master", "Bac", "Bac", "Master", "Master", NA, "Bac", "Bac")
sexe <- c("H", "H", "F", "H", "H", "H", "F", "H", "H", "H") |> factor()# A tibble: 10 × 4
height weight prog sexe
<dbl> <dbl> <chr> <fct>
1 167 86 Bac H
2 192 74 Bac H
3 173 83 Master F
4 174 50 Bac H
5 172 78 Bac H
6 167 66 Master H
7 171 66 Master F
8 185 51 <NA> H
9 163 50 Bac H
10 170 55 Bac H L’affichage des tibbles est aussi similaire à celui des dataframes avec toutefois quelques particularités, notamment :
- La première ligne montre la nature de l’objet (tibble) et ces dimensions (ici 10 x 4, pur 10 lignes et 4 colonnes).
- Sous les noms de colonnes, les types de données sont indiqués en abrégé.
- Le type de chaque variable/colonne visible est décrit, en abrégé, juste après les noms de colonnes.
- Par défaut, les tibbles affichent uniquement les 10 premières lignes et autant de colonnes que la largeur de la console le permet, avec un message indiquant les lignes ou colonnes non affichées.
Aussi, il est possible de créer un tible ligne par ligne à l’aide de la fonction tribble().
tribble(
~height, ~weight, ~prog, ~sexe,
67, 86, "Bac+2", "H",
192, 74, "Bac", "H",
173, 83, "Master", "F"
)# A tibble: 3 × 4
height weight prog sexe
<dbl> <dbl> <chr> <chr>
1 67 86 Bac+2 H
2 192 74 Bac H
3 173 83 Master F Malgré ces différences, un tibble et un dataframe se manipulent exactement de la même manière. On peut donc appliquer sur un tibble tout ce que l’on a appris avec les dataframes. Il est par ailleurs possible de transformer un tibble en dataframe, et vice versa.
'data.frame': 10 obs. of 4 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac" "Bac" "Master" "Bac" ...
$ sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2tibble [10 × 4] (S3: tbl_df/tbl/data.frame)
$ height: num [1:10] 167 192 173 174 172 167 171 185 163 170
$ weight: num [1:10] 86 74 83 50 78 66 66 51 50 55
$ prog : chr [1:10] "Bac" "Bac" "Master" "Bac" ...
$ sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 27.4.2 Data Tables
Le package data.table a été conçu pour surmonter les limites des dataframes traditionnels, en particulier lorsqu’il s’agit de réaliser des calculs intensifs ou de manipuler de très grands volumes de données.
+ Création et affichage
height weight prog sexe
<num> <num> <char> <fctr>
1: 167 86 Bac H
2: 192 74 Bac H
3: 173 83 Master F
4: 174 50 Bac H
5: 172 78 Bac H
6: 167 66 Master H
7: 171 66 Master F
8: 185 51 <NA> H
9: 163 50 Bac H
10: 170 55 Bac HClasses 'data.table' and 'data.frame': 10 obs. of 4 variables:
$ height: num 167 192 173 174 172 167 171 185 163 170
$ weight: num 86 74 83 50 78 66 66 51 50 55
$ prog : chr "Bac" "Bac" "Master" "Bac" ...
$ sexe : Factor w/ 2 levels "F","H": 2 2 1 2 2 2 1 2 2 2
- attr(*, ".internal.selfref")=<externalptr> + Syntaxe et opérations fondamentales
Avec data.table, il est possible d’imbriquer des opérations de filtrage, de sélection et d’agrégation dans une seule instruction, grâce au syntaxe :
ipour la sélection et filtrage sur les lignes.jpour la sélection et transformation sur les colonnes.bypour regroupement et agrégation.
L’expression dt[i, j, by] est évaluée dans l’ordre suivant : d’abord, les lignes sont sélectionnées (ou filtrées) selon i; ensuite, les données filtrées sont regroupées selon by; enfin, les opérations spécifiées dans j sont appliquées sur les données ainsi filtrées et groupées. Il est également utile de noter qu’il n’est pas nécessaire d’utiliser le symbole $ à l’intérieur des crochets [...], ce qui permet d’écrire un code bien plus lisible.
Examinons quelques exemples :
height weight prog sexe
<num> <num> <char> <fctr>
1: 167 86 Bac H
2: 192 74 Bac H height weight prog sexe
<num> <num> <char> <fctr>
1: 167 86 Bac H
2: 192 74 Bac H
3: 173 83 Master F
4: 172 78 Bac H height weight
<num> <num>
1: 167 86
2: 192 74
3: 173 83
4: 174 50
5: 172 78
6: 167 66
7: 171 66
8: 185 51
9: 163 50
10: 170 55 weight prog sexe
<num> <char> <fctr>
1: 86 Bac H
2: 74 Bac H
3: 83 Master F
4: 50 Bac H
5: 78 Bac H
6: 66 Master H
7: 66 Master F
8: 51 <NA> H
9: 50 Bac H
10: 55 Bac H weight prog sexe
<num> <char> <fctr>
1: 86 Bac H
2: 74 Bac H
3: 83 Master F
4: 78 Bac HL’équivalent du code ci-dessus en R de base est le suivant :
df[c(1, 2), ]
dt |> subset(weight > median(weight))
df[c(1, 2)]
df[c("weight", "prog", "sexe")]
df[c("weight", "prog", "sexe")] |> subset(weight > median(weight))La composante j ne sert pas uniquement à sélectionner des colonnes, mais permet aussi, contrairement aux dataframes, d’effectuer des calculs sur celles-ci.
[1] 65.9 mean median
<num> <num>
1: 65.9 66Il également possible de créer de nouvelles variables ou de transformer celles existantes via l’opérateur :=
height weight prog sexe V
<num> <num> <char> <fctr> <num>
1: 167 86 Bac H 6
2: 192 74 Bac H 6
3: 173 83 Master F 6
4: 174 50 Bac H 6
5: 172 78 Bac H 6
6: 167 66 Master H 6
7: 171 66 Master F 6
8: 185 51 <NA> H 6
9: 163 50 Bac H 6
10: 170 55 Bac H 6 height weight prog sexe imc
<num> <num> <char> <fctr> <num>
1: 167 86 Bac H 30.8
2: 192 74 Bac H 20.1
3: 173 83 Master F 27.7
4: 174 50 Bac H 16.5
5: 172 78 Bac H 26.4
6: 167 66 Master H 23.7
7: 171 66 Master F 22.6
8: 185 51 <NA> H 14.9
9: 163 50 Bac H 18.8
10: 170 55 Bac H 19.0À noter que := ne renvoie aucun résultat, mais agit directement sur les données en mémoire (sans créer de copie). Cette approche est beaucoup plus rapide et efficace, en particulier lorsqu’on travaille avec de très grands volumes de données.
Les opérations sur les colonnes peuvent être effectuées par groupe à l’aide de l’argument by
# Version 1 : Crée un nouveau tableau avec le résultat des calculs
dt[, .(mean = mean(weight)), by = .(sexe, prog)] sexe prog mean
<fctr> <char> <num>
1: H Bac 65.5
2: F Master 74.5
3: H Master 66.0
4: H <NA> 51.0 height weight prog sexe imc mean
<num> <num> <char> <fctr> <num> <num>
1: 167 86 Bac H 30.8 65.5
2: 192 74 Bac H 20.1 65.5
3: 173 83 Master F 27.7 74.5
4: 174 50 Bac H 16.5 65.5
5: 172 78 Bac H 26.4 65.5
6: 167 66 Master H 23.7 66.0
7: 171 66 Master F 22.6 74.5
8: 185 51 <NA> H 14.9 51.0
9: 163 50 Bac H 18.8 65.5
10: 170 55 Bac H 19.0 65.5Dans by on peut aussi fournir une ou plusieurs conditions logiques :
weight > median(weight) mean
<lgcl> <num>
1: TRUE 80.2
2: FALSE 56.3En plus, il est possible d’enchaîner plusieurs opérations en utilisant les crochets [...] les uns après les autres selon la syntaxe
Chaque opération dans [] prend le data.table provenant de l’opération précédente et lui applique une nouvelle transformation. Le résultat final est le cumul de toutes ces transformations appliquées séquentiellement. Voici un exemple.
dt[
height > median(height), .(weight, prog, sexe)
][
, .(mean = mean(weight)), by = sexe
][
order(sexe)] sexe mean
<fctr> <num>
1: F 83.0
2: H 63.2Ce qui peut se traduire par
- Partir de
dt, filtrer les lignes pour ne garder que les individus dontheightest supérieure à sa médiane. On ne conserve que les colonnesweight,progetsexe. - Partir des données obtenues en (1), calculer le poids moyen par
sexeet enregistrer le résulta dansmean.w. - Partir des données obtenues en (2), trier les lignes par ordre alphabétique du
sexe.
Le package data.table propose également un certain nombre d’opérateurs spéciaux (appelés aussi variables), comme par exemple .N, qui sert simplement à compter le nombre de lignes.
[1] 10 sexe prog N
<fctr> <char> <int>
1: H Bac 6
2: F Master 2
3: H Master 1
4: H <NA> 1 sexe prog mean effectif
<fctr> <char> <num> <int>
1: H Bac 65.5 6
2: F Master 74.5 2
3: H Master 66.0 1
4: H <NA> 51.0 1Ceci n’est qu’une introduction aux éléments de base. Pour en savoir plus, vous pouvez consulter la documentation officielle : https://rdatatable.gitlab.io/data.table/.
7.5 Sources de données
Une analyse de données débute typiquement par l’acquisition de données qui peut se faire de différentes manières et sous différentes formes. Nous allons survoler (très brièvement) quelques façons de réaliser cette première étape.
Packages R
De très nombreuses packages viennent avec plusieurs jeux de données accessibles une fois le package chargé. C’est le cas, par exemple, du package de base datasets qui est automatiquement installé et chargé par R.
Pour avoir une liste complète de tous les données disponibles dans les packages chargés dans la session R en cours, tapez :
La sortie de cette commande varie en fonction des packages chargés. Par exemple, après avoir charger le package MASS, voici la sortie qu’on obtient
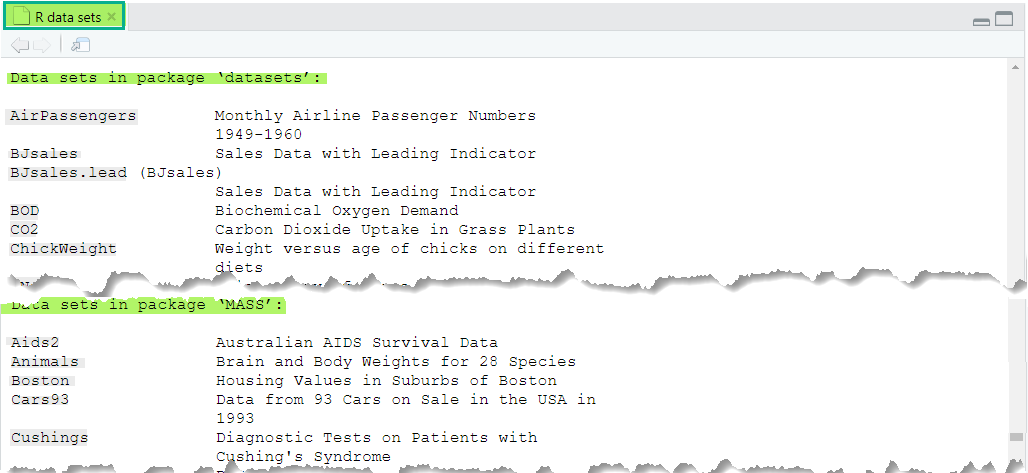
Il s’agit des noms suivis par une brève description pour chaque jeu de données. Si vous voulez voir uniquement les données disponible dans MASS, tapez
Parmi les noms affichés il y a, par exemple,Animals. Si vous tapez ce nom dans la console, vous verrez le contenu de ce dataframe. Pour obtenir une description de ces données, tapez
Fichiers externes
Très souvent les données à analyser sont disponibles dans un fichier externe (local ou sur le web) sous divers formats : texte délimités (dont .txt, .csv, .dsv, etc.), JSON, EXCEL, etc. Nous aborderons ici uniqument le format texte qui est le plus rencontré en pratique.
Pour la suite de la démonstration, nous allons utiliser le fichier Med.csv qu’on vous invite à télécharger et sauvegarder sur votre machine. Voici un aperçu du contenu de ce fichier.
subject sex condition before after change
1 F placebo 10.1 6.9 -3.2
2 F placebo 6.3 4.2 -2.1
3 M aspirin 12.4 6.3 -6.1
4 F placebo 8.1 6.1 -2.0
5 M aspirin 15.2 9.9 -5.3
6 F aspirin 10.9 7.0 -3.9Pour importer ces données, nous allons utiliser la fonction read.csv(). Cette fonction accepte beaucoup d’arguments qui permettent de s’adapter à la nature de fichier à importer; voir le Help de cette fonction pour plus de détails. Parmi ces arguments il y a : (1) header : valeur logique (TRUE ou FALSE) pour la présence ou non d’un en-tête avec les noms des variables (par défaut TRUE), (2) sep : la maniéré dont les champs (variables) sont séparés (par défaut une virgule), et (3) dec: le séparateur décimal (par défaut un point).
Le seul argument obligatoire est le chemin d’accès au fichier à lire. Mais, comme déjà expliqué, il n’est pas nécessaire de spécifier le chemin complet si ce le fichier à lire se trouve dans votre Working directory. Si c’est le ce cas, pour charger “Med.csv” dans R, il suffit de taper
De là, on peut utiliser l’objet Med comme tout autre dataframe en R.
RStudio fournit une interface graphique simple pour faciliter l’import d’un fichier. Pour cela, il suffit d’aller dans le menu File > Import Dataset > From Text (base), ou via l’onglet Environment, et indiquer l’emplacement du fichier et ces caractéristiques.
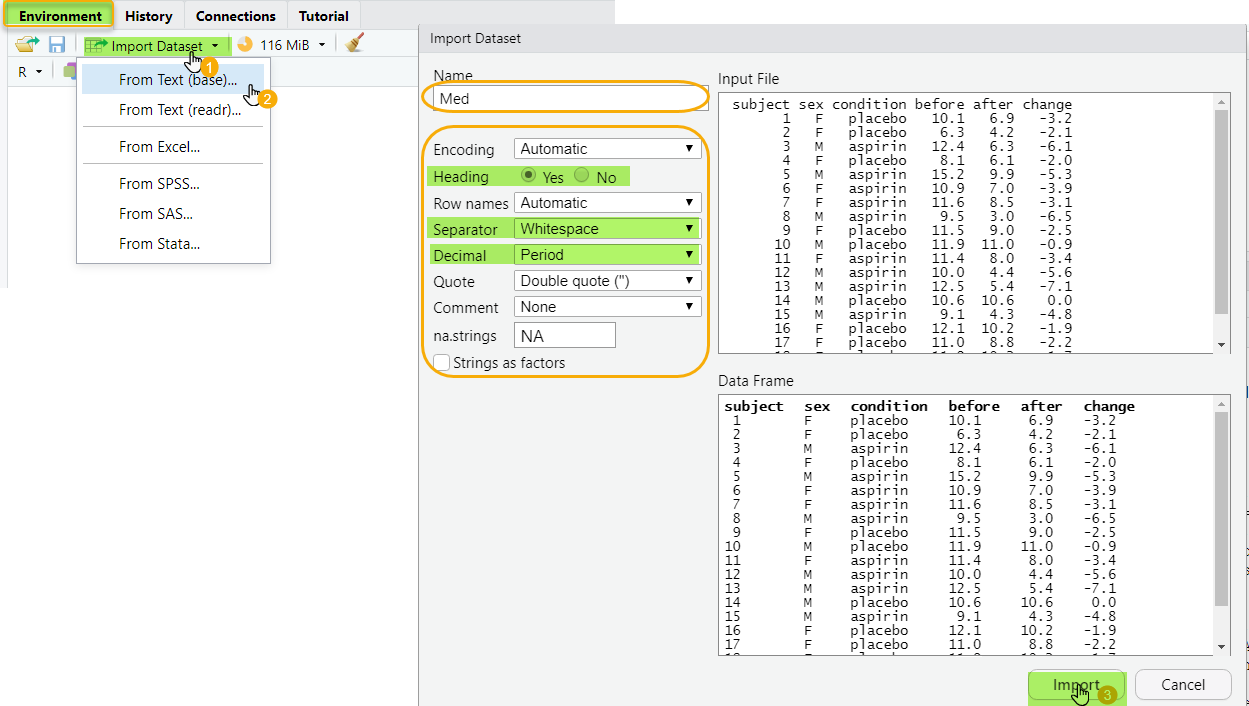
Le code généré et les données importées sont automatiquement affichés.
On peut aussi exporter des données à partir de R vers un fichier texte. Pour cela, il suffit d’utiliser la fonction write.csv().